搜索


近日,新澳网门票官方网站新澳网门票官方网站陈海峰教授课题组在J. Chem. Theory Comput. 期刊在线发表题为“Well-balanced Force Field ff03CMAP for Folded and Disordered Proteins”的研究成果,提供了一种新的高效而平衡的、能够同时模拟结构蛋白和天然无规蛋白的分子力场。硕士生张阳鹏为第一作者,陈海峰教授为通讯作者。
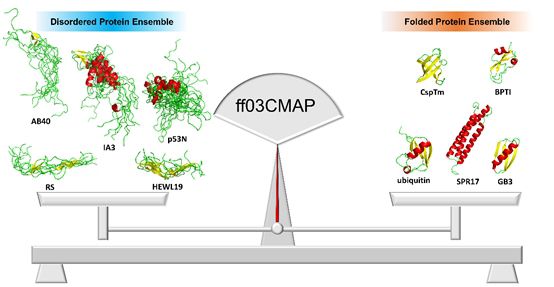
蛋白质是生命的物质基础,蛋白质有结构蛋白和天然无规蛋白两种类别。天然无规蛋白是一类在生理条件下没有稳定三级结构的蛋白质,这类蛋白在真核生物蛋白质组中的含量超过40%,与肿瘤、心血管疾病、神经退行性疾病以及糖尿病等复杂疾病的发生发展密切相关。结构蛋白的序列结构功能范式已经被大家广泛接受,但天然无规蛋白的序列无规功能范式迫切需要建立。与结构蛋白相比,天然无规蛋白构象多样性等特点导致难以用X-ray、NMR等传统实验方法来研究。分子动力学模拟是研究天然无规蛋白的序列无规功能范式的重要方法,分子力场是分子动力学模拟的基础,但现有的分子力场尚不能同时模拟结构蛋白与天然无规蛋白。因而,依赖于高性能计算的分子动力学模拟在天然无规蛋白的研究中发挥着越来越重要的作用。
由此,陈海峰教授课题组在ff03分子力场的基础上,通过添加基于能量的矫正项,发展了一种平衡而高效的分子力场ff03CMAP,为天然无规蛋白的序列无规功能范式的建立奠定了坚实基础。通过对短肽、天然无规蛋白、结构蛋白以及快速折叠蛋白等的大量测试结果表明,该分子力场模拟的化学位移、J-耦合,序参数以及残基偶极耦合都与NMR试验观察高度一致,同时比其它分子力场的模拟结果更加准确。
该分子力场的发布对于揭示天然无规蛋白的序列无规功能范式以及基于天然无规蛋白的复杂疾病发生发展的分子机制都具有重要的意义。
该研究获得国家重点研发计划以及国家自然科学基金面上项目的资助。
论文链接:https://pubs.acs.org/doi/10.1021/acs.jctc.9b00623



 首页
首页

 学院概况
学院概况

 师资队伍
师资队伍

 人才培养
人才培养

 招生就业
招生就业

 科学研究
科学研究

 平台基地
平台基地

 党群工作
党群工作

 校友之家
校友之家

 安全工作
安全工作

 网上办事
网上办事

 当前位置:
当前位置: